Трансформеры — большой тренд в компьютерном зрении. Недавно я сделал обзор некоторых удивительных достижений. На этот раз я буду использовать свою повторную реализацию модели на основе трансформатора для 3D-сегментации. В частности, я буду использовать знаменитый преобразователь UNITR и попытаюсь посмотреть, работает ли он наравне с классическим UNET. блокнот доступен.
ЮНЕТР является первой успешной архитектурой-трансформером для сегментации 3D-медицинских изображений. В этом сообщении блога я попытаюсь сопоставить результаты модели UNET с набором данных BRATS, который содержит трехмерные МРТ-изображения мозга. Вот общий обзор UNITR, которому мы будем обучать в этом руководстве:
Источник: UNITR: Transformers for 3D Medical Image Segmentation, Hatamizadeh et al.
Чтобы проверить свою реализацию, я использовал существующий учебник на наборе данных сегментации 3D МРТ. Таким образом, я должен отдать должное удивительной библиотеке Nvidia с открытым исходным кодом под названием МОНАИ за предоставленный начальный учебник, который я изменил в образовательных целях. Если вы увлекаетесь медицинской визуализацией, обязательно ознакомьтесь с этой замечательной библиотекой и ее учебными пособиями.
Давайте сначала посмотрим данные!
Обновление: выпуск книги! Узнайте о «Глубоком обучении в производственной среде», чтобы предоставлять свои модели машинного обучения миллионам пользователей.
Набор данных BRATS
БРАТС представляет собой мультимодальный крупномасштабный набор данных трехмерных изображений. Он содержит 4 3D-тома МРТ-изображений, снятых при различных условиях и настройках. Вот пример набора данных. Важно видеть, что аннотируется только опухоль. Это усложняет такие вещи, как сегментация, поскольку модель должна быть локализована на опухоли.
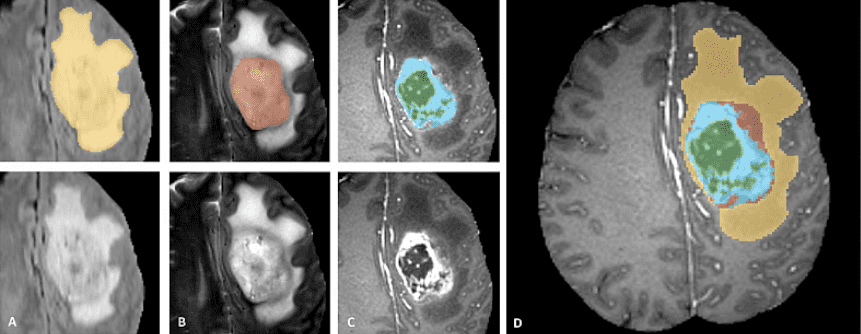
Официальное тизерное изображение данных с веб-сайта завершения BRATS.
Патчи изображения изображают категории опухолей следующим образом (слева направо):
-
Отек: Вся опухоль (желтая) обычно видна на МРТ-изображении T2-FLAIR.
-
Неармирующее твердое ядро: Ядро опухоли (красное) видно на Т2 МРТ.
-
усиливающая опухоль структуры (светло-голубой). Обычно виден в T1Gd, вокруг некротическое ядро (зеленый).
-
Сегментации объединяются для создания окончательных меток набора данных.
С MONAIзагружая набор данных из соревнования по медицинской визуализации в десятиборье становится тривиальным.
Загрузка данных с помощью MONAI и преобразования
Используя DecathlonDataset класса библиотеки MONAI можно загрузить любой из 10 доступных наборов данных с веб-сайта. Мы будем использовать Task01_BrainTumour в нашем случае.
cache_num = 8
from monai.apps import DecathlonDataset
train_ds = DecathlonDataset(
root_dir=root_dir,
task="Task01_BrainTumour",
transform=train_transform,
section="training",
download=True,
num_workers=4,
cache_num=cache_num,
)
train_loader = DataLoader(train_ds, batch_size=2, shuffle=True, num_workers=2)
val_ds = DecathlonDataset(
root_dir=root_dir,
task="Task01_BrainTumour",
transform=val_transform,
section="validation",
download=False,
num_workers=4,
cache_num=cache_num,
)
val_loader = DataLoader(val_ds, batch_size=2, shuffle=False, num_workers=2)
Импорт и вспомогательные функции можно найти в блокнот. Что здесь важно, так это конвейер преобразования, который, я гарантирую, непрост в 3D-изображениях. MONAI предоставляет некоторые функции для создания быстрого конвейера для целей этого руководства. Такие детали, как ориентация изображения, намеренно опущены в учебнике.
Вкратце, мы передискретизируем наши изображения до размера вокселя 1,5, 1,5 и 2,0 мм в каждом измерении. После этого мы берем случайные 3D-подобъемы размеров 128, 128, 64. Это, конечно, необходимо применить как к входному изображению, так и к маске сегментации.
Затем применяется пара дополнений, таких как случайное переворачивание первой оси и изменение масштаба интенсивности (дрожание).
Класс ConvertToMultiChannelBasedOnBratsClassesd приводит метки к нужному нам формату.
from monai.transforms import (
Activations,
AsChannelFirstd,
AsDiscrete,
CenterSpatialCropd,
Compose,
LoadImaged,
MapTransform,
NormalizeIntensityd,
Orientationd,
RandFlipd,
RandScaleIntensityd,
RandShiftIntensityd,
RandSpatialCropd,
Spacingd,
ToTensord,
)
roi_size=(128, 128, 64)
pixdim=(1.5, 1.5, 2.0)
class ConvertToMultiChannelBasedOnBratsClassesd(MapTransform):
"""
Convert labels to multi channels based on brats classes:
label 1 is the peritumoral edema
label 2 is the GD-enhancing tumor
label 3 is the necrotic and non-enhancing tumor core
The possible classes are TC (Tumor core), WT (Whole tumor)
and ET (Enhancing tumor).
"""
def __call__(self, data):
d = dict(data)
for key in self.keys:
result = ()
result.append(np.logical_or(d(key) == 2, d(key) == 3))
result.append(
np.logical_or(
np.logical_or(d(key) == 2, d(key) == 3), d(key) == 1
)
)
result.append(d(key) == 2)
d(key) = np.stack(result, axis=0).astype(np.float32)
return d
train_transform = Compose(
(
LoadImaged(keys=("image", "label")),
AsChannelFirstd(keys="image"),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Spacingd(
keys=("image", "label"),
pixdim=pixdim,
mode=("bilinear", "nearest"),
),
Orientationd(keys=("image", "label"), axcodes="RAS"),
RandSpatialCropd(
keys=("image", "label"), roi_size=roi_size, random_size=False),
RandFlipd(keys=("image", "label"), prob=0.5, spatial_axis=0),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
RandScaleIntensityd(keys="image", factors=0.1, prob=0.5),
RandShiftIntensityd(keys="image", offsets=0.1, prob=0.5),
ToTensord(keys=("image", "label")),
)
)
val_transform = Compose(
(
LoadImaged(keys=("image", "label")),
AsChannelFirstd(keys="image"),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Spacingd(
keys=("image", "label"),
pixdim=pixdim,
mode=("bilinear", "nearest"),
),
Orientationd(keys=("image", "label"), axcodes="RAS"),
CenterSpatialCropd(keys=("image", "label"), roi_size=roi_size),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
ToTensord(keys=("image", "label")),
)
)
Всегда лучше увидеть конвейер в действии, визуализировав несколько срезов всех модальностей. Ниже приведен пример данных о наших поездах:
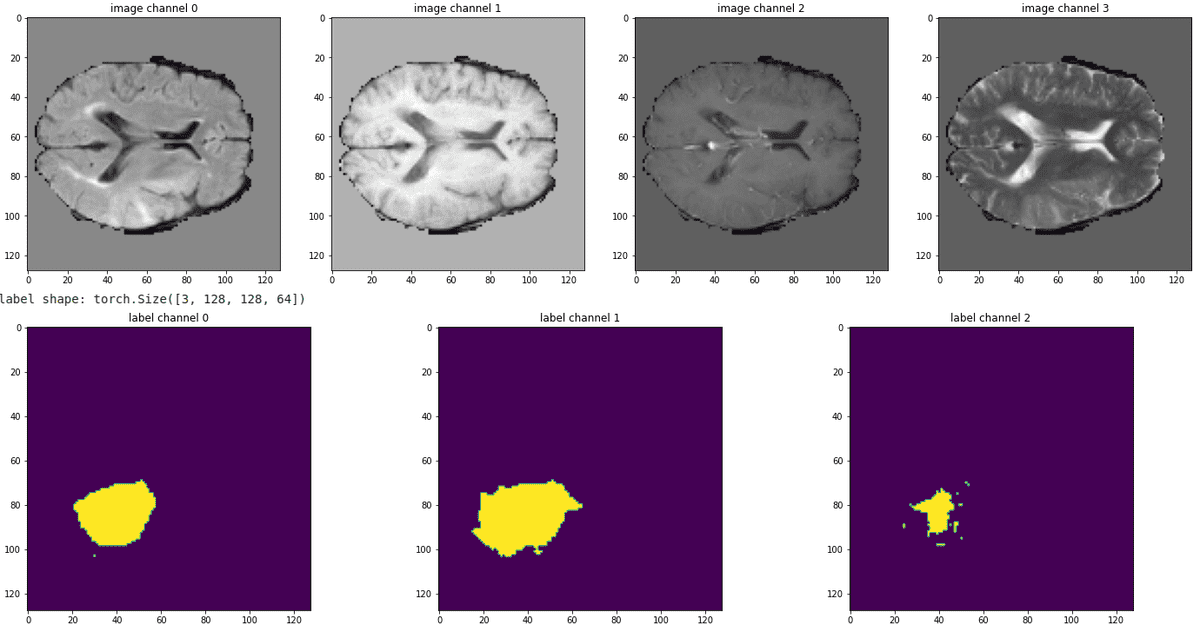
Источник: изображение автора на основе блокнота.
Можно заметить, что опухоль нет взаимоисключающий. В связи с этим мы ожидаем, что усиление опухолевых и некротических клеток (крайняя правая карта сегментации) будет наиболее трудным для прогнозирования.
Конвейер данных и преобразования теперь настроен. Рассмотрим подробнее архитектуру модели.
Узнайте больше об искусственном интеллекте, применяемом в приложениях для обработки медицинских изображений, из хорошо структурированного курса. ИИ для медицины предлагает Курсера.
Архитектура ЮНЕТР
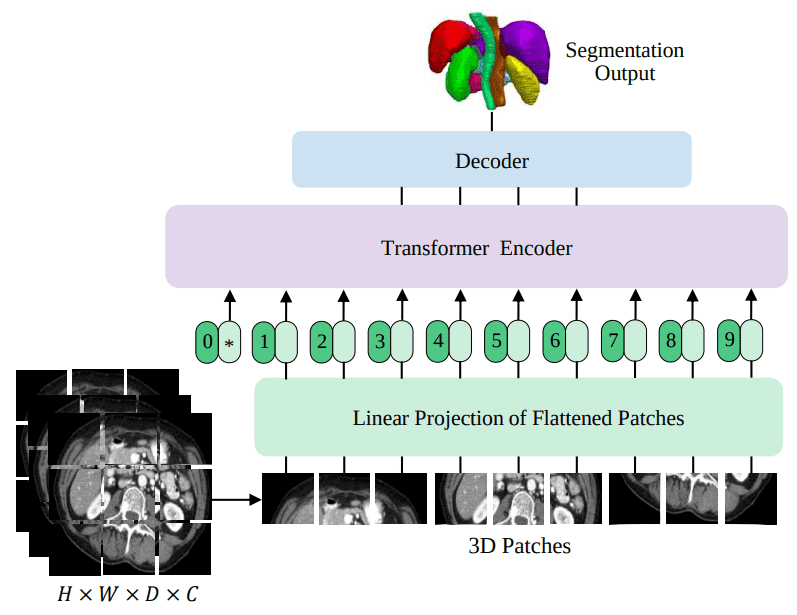
Вот архитектура модели, которая включает преобразователи в печально известную архитектуру UNET:
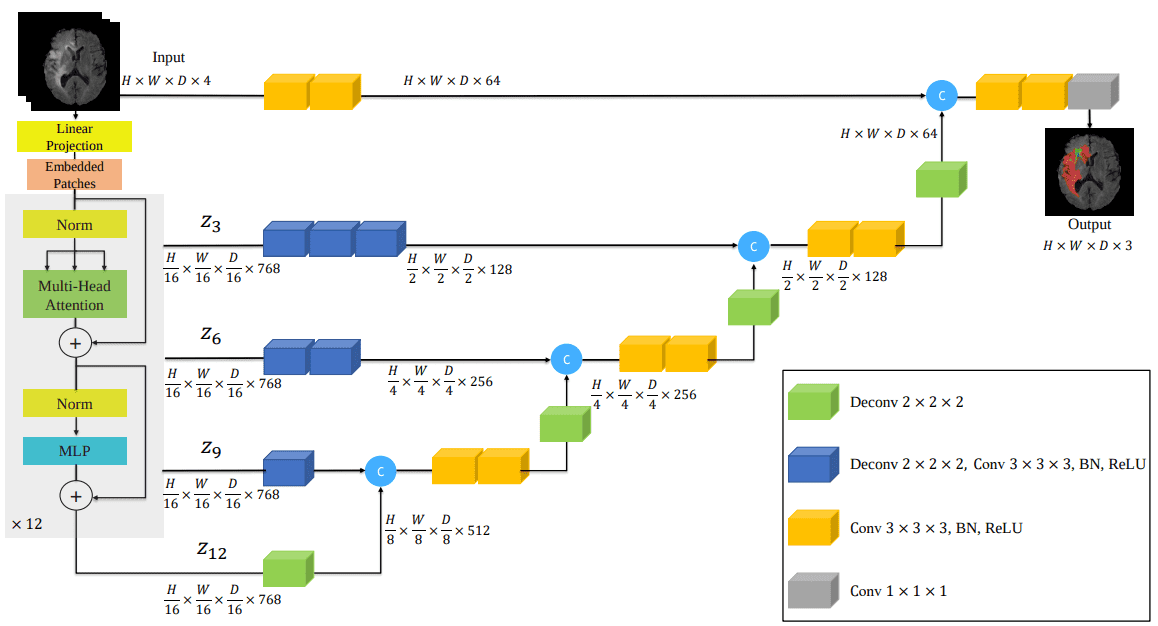
Источник: UNITR: Transformers for 3D Medical Image Segmentation, Hatamizadeh et al.
Интересно, что эту модель я начал реализовывать как на бумажном рисунке, изображенном выше. Позже я обнаружил, что это уже реализовано в MONAI. После проверки их кода я обнаружил, что отсутствуют важные детали. Вывод: не доверяйте архитектурным изображениям, они не включают всю историю о том, как реализовать статью. Чтобы увидеть код реализации, ознакомьтесь с моей реализацией в само-внимание-резюме библиотека.
Теперь я наконец могу использовать свою реализацию UNITR. Я создал небольшую библиотеку, которая реализует несколько блоков самоконтроля для компьютерного зрения и упаковывает их в устанавливаемый pip-пакет. Итак, теперь мне нужно только установить пакет pip, содержащий модель, и вуаля:
$ pip install self-attention-cv==1.2.3
Чтобы инициализировать модель, нам нужно предоставить размер тома, модальности входных изображений, количество меток (output_dim) и несколько вещей, касающихся преобразователя зрения. Примеры включают размер встраивания патча, размер патча, количество головок, тип нормализации и т. д.
from self_attention_cv import UNETR
device = torch.device("cuda:0")
num_heads = 10
embed_dim= 512
model = UNETR(img_shape=tuple(roi_size), input_dim=4, output_dim=3,
embed_dim=embed_dim, patch_size=16, num_heads=num_heads,
ext_layers=(3, 6, 9, 12), norm='instance',
base_filters=16,
dim_linear_block=2048).to(device)
Я до сих пор не уверен, почему нормализация экземпляров очень хорошо работает с UNET и мультимодельными наборами данных, но это так! Дело в том, что у нас есть готовая к обучению модель с 49,7 миллионами параметров.
Мы будем использовать DICE потеря в сочетании с кросс-энтропией, и сделайте простой тренировочный цикл:
import torch.nn as nn
from monai.losses import DiceLoss, DiceCELoss
loss_function = DiceCELoss(to_onehot_y=False, sigmoid=True)
optimizer = torch.optim.AdamW(model.parameters(), lr=1e-4, weight_decay=1e-5)
max_epochs = 180
val_interval = 5
best_metric = -1
best_metric_epoch = -1
epoch_loss_values = ()
for epoch in range(max_epochs):
print(f"epoch {epoch + 1}/{max_epochs}")
model.train()
epoch_loss = 0
step = 0
for batch_data in train_loader:
step += 1
inputs, labels = (
batch_data("image").to(device),
batch_data("label").to(device),
)
optimizer.zero_grad()
outputs = model(inputs)
loss = loss_function(outputs, labels)
loss.backward()
optimizer.step()
epoch_loss += loss.item()
epoch_loss /= step
epoch_loss_values.append(epoch_loss)
print(f"epoch {epoch + 1} average loss: {epoch_loss:.4f}")
Базовое сравнение: UNET
Тем не менее, самый большой вопрос здесь заключается в том, насколько хороша эта модель. По этой причине нам нужна сильная основа! Что может быть лучше хорошо сконфигурированного UNET, который использовался в начальном руководстве?
Я также сравнил свою реализацию с реализацией UNITR от MONAI. Почему? Потому что не будет никакого смысла, если я сравняюсь по производительности с базовым уровнем UNET и все равно уступлю официальной реализации. В конце концов, я изменил свой код, чтобы отразить архитектурные изменения официального кода. И действительно, я увидел огромный прирост производительности по сравнению с упрощенной реализацией на бумаге.
from monai.networks.nets import UNet
model = UNet(
dimensions=3,
in_channels=4,
out_channels=3,
channels=(16, 32, 64, 128, 256),
strides=(2, 2, 2, 2),
num_res_units=2,
).to(device)
Давайте сначала посмотрим число:
| Модель | эпохи | Средний коэффициент DICE. |
| UNET (базовый уровень) | 170 | 76,6 % |
| UNITR (самостоятельное внимание) | 180 | 76,9 % |
| ЮНЕТР (МОНАИ) | 180 | 76,1 % |
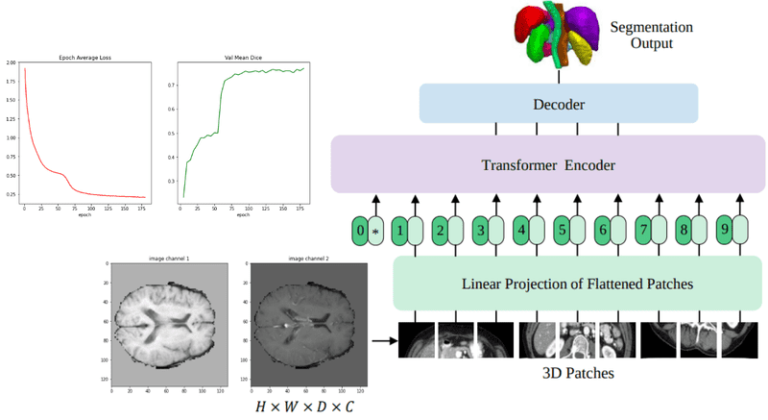
Чтобы отслеживать обучение, мы измеряем потери при обучении как по потерям в кубиках, так и по кросс-энтропии. Мы также сообщаем коэффициенты кубиков для 3 меток (каналов), а именно ядра опухоли (TC), всей опухоли (WT) и усиливающейся опухоли (EC).
Ниже вы можете увидеть эти показатели во время обучения:
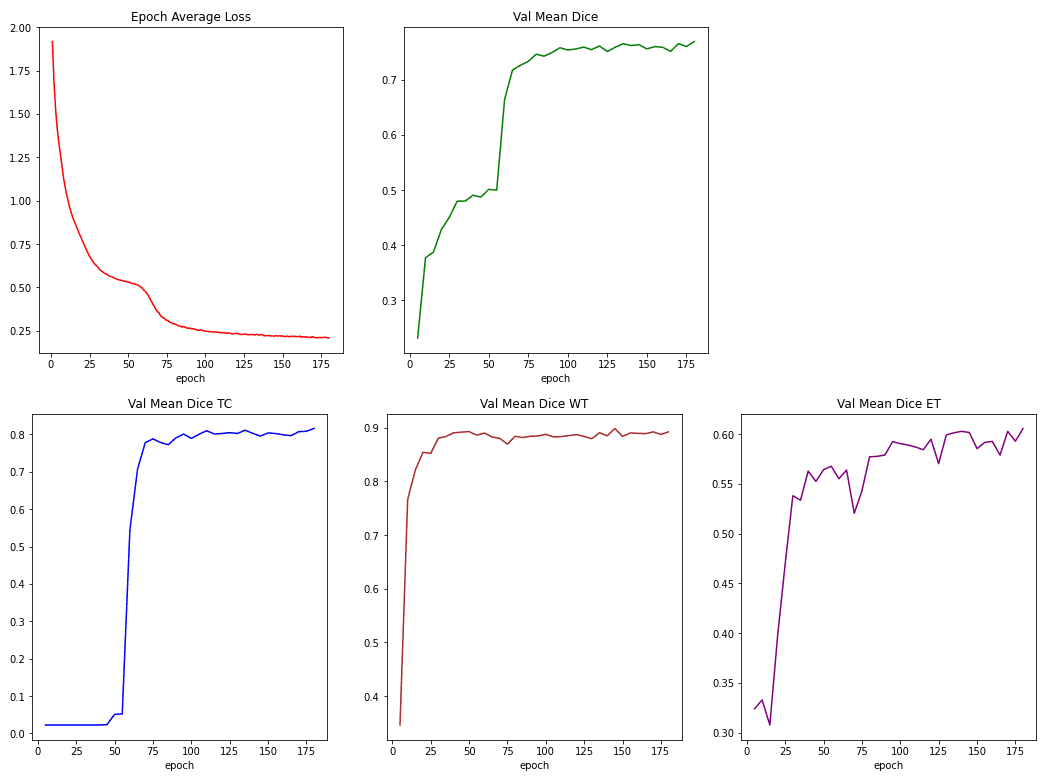
Источник: изображение автора на основе блокнота.
Наконец, можно увидеть результаты, сравнив выходную карту сегментации с реальной правдой:
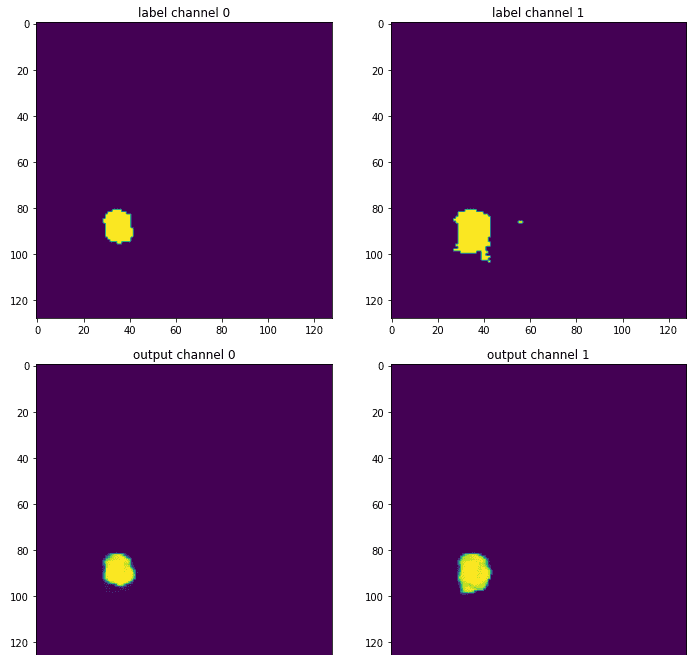
Источник: изображение автора на основе блокнота.
Канал некротической зоны опущен, потому что на этом конкретном срезе эта метка почти не встречается. Эта иллюстрация является лишь средним фрагментом карты 3D-сегментации, так что это, конечно, не вся картина. Тем не менее, это дает вам представление о том, как обученная модель обеспечивает более сглаженную версию исходной метки, которая была прокомментирована опытным рентгенологом. Потому что, как всегда, нейронные сети любят гладкие пространства оптимизации.
Заключение и опасения
Я еще не уверен в эффективности трансформеров в трехмерной медицинской визуализации. Я считаю, что более продвинутые методы и другие вклады последуют. Тем не менее, я признаю, что это первая интересная работа, которая бросает вызов хорошо сконфигурированной архитектуре UNET, которая является предпочтительным вариантом в этих задачах.
Исходя из вышеприведенного анализа, я считаю важным подчеркнуть также, что наиболее важным аспектом для получения хорошей производительности, здесь коэффициентом Dice, являются конвейеры предварительной обработки и преобразования данных. Именно поэтому я вижу ограниченные инновации в мире медицинской визуализации с точки зрения моделирования машинного обучения и более многообещающую работу по оптимизации обработки данных. Само по себе это не вызывает никаких проблем, но вызывает у меня большие подозрения, когда выходит новая статья и заявляется о новой архитектуре. Потому что сравнения часто несправедливы в нишевых областях, над которыми мне довелось работать, таких как медицинская визуализация.
Как всегда, спасибо за ваш интерес к ИИ и следите за обновлениями. Мы с гордостью делимся с вами нашей книгой «Глубокое обучение в производстве», в которой вы узнаете, как запустить вашу модель в производство и масштабировать ее. Поддержка сообщества (например, совместное использование социальных сетей) всегда приветствуется.
* Раскрытие информации: обратите внимание, что некоторые из приведенных выше ссылок могут быть партнерскими ссылками, и мы без дополнительных затрат для вас получим комиссию, если вы решите совершить покупку после перехода по ссылке.