В этом посте мы демонстрируем, как эффективно настроить современную модель белкового языка (pLM) для прогнозирования субклеточной локализации белка с помощью Amazon SageMaker.
Белки — это молекулярные механизмы организма, отвечающие за все: от движения мышц до реакции на инфекции. Несмотря на такое разнообразие, все белки состоят из повторяющихся цепочек молекул, называемых аминокислотами. Геном человека кодирует 20 стандартных аминокислот, каждая из которых имеет несколько разную химическую структуру. Они могут быть представлены буквами алфавита, что позволяет нам анализировать и исследовать белки в виде текстовой строки. Огромное возможное количество белковых последовательностей и структур — вот что дает белкам широкое разнообразие применений.

Белки также играют ключевую роль в разработке лекарств, будучи не только потенциальными мишенями, но и терапевтическими средствами. Как показано в следующей таблице, многие из самых продаваемых лекарств в 2022 году представляли собой либо белки (особенно антитела), либо другие молекулы, такие как мРНК, трансформированные в белки в организме. Из-за этого многим исследователям в области медико-биологических наук необходимо быстрее, дешевле и точнее отвечать на вопросы о белках.
| Имя | Производитель | Мировые продажи в 2022 году (миллиарды долларов США) | Показания |
| Комирнаты | Пфайзер/БиоНТек | $40,8 | COVID-19 |
| Спикевакс | Модерна | $21,8 | COVID-19 |
| Хумира | ЭббВи | $21,6 | Артрит, болезнь Крона и другие. |
| Кейтруда | Мерк | $21,0 | Различные виды рака |
Источник данных: Уркарт, Л. Топ компаний и лекарств по продажам в 2022 году. Nature Reviews Drug Discovery 22, 260–260 (2023).
Поскольку мы можем представлять белки как последовательности символов, мы можем анализировать их, используя методы, изначально разработанные для письменного языка. Сюда входят большие языковые модели (LLM), предварительно обученные на огромных наборах данных, которые затем можно адаптировать для конкретных задач, таких как суммирование текста или чат-боты. Точно так же pLM предварительно обучаются на больших базах данных последовательностей белков с использованием немаркированного обучения с самоконтролем. Мы можем адаптировать их для прогнозирования таких вещей, как трехмерная структура белка или то, как он может взаимодействовать с другими молекулами. Исследователи даже использовали pLM для разработки новых белков с нуля. Эти инструменты не заменяют научный опыт человека, но у них есть потенциал ускорить доклинические разработки и планирование исследований.
Одной из проблем этих моделей является их размер. Как LLM, так и PLM выросли на порядки за последние несколько лет, как показано на следующем рисунке. Это означает, что на их обучение до достаточной точности может уйти много времени. Это также означает, что вам необходимо использовать оборудование, особенно графические процессоры, с большим объемом памяти для хранения параметров модели.

Длительное время обучения плюс большие экземпляры обозначают высокую стоимость, что может сделать эту работу недоступной для многих исследователей. Например, в 2023 году Исследовательская команда описал обучение pLM со 100 миллиардами параметров на 768 графических процессорах A100 в течение 164 дней! К счастью, во многих случаях мы можем сэкономить время и ресурсы, адаптировав существующую PLM под нашу конкретную задачу. Эта техника называется тонкая настройкаа также позволяет нам заимствовать передовые инструменты из других типов языкового моделирования.
Обзор решения
Конкретная проблема, которую мы рассматриваем в этом посте, заключается в субклеточная локализация: Учитывая последовательность белка, можем ли мы построить модель, которая сможет предсказать, живет ли он снаружи (клеточная мембрана) или внутри клетки? Это важная информация, которая может помочь нам понять функцию и то, станет ли она хорошей мишенью для лекарств.
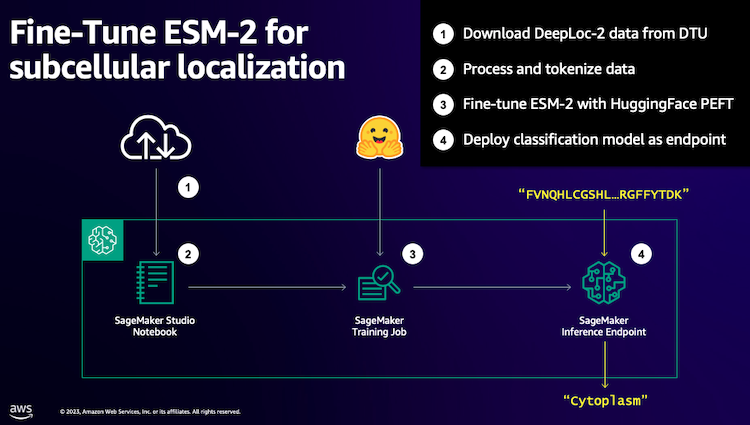
Начнем с загрузки общедоступного набора данных с помощью Amazon SageMaker Studio. Затем мы используем SageMaker для точной настройки модели белкового языка ESM-2, используя эффективный метод обучения. Наконец, мы развертываем модель в качестве конечной точки вывода в реальном времени и используем ее для тестирования некоторых известных белков. Следующая диаграмма иллюстрирует этот рабочий процесс.

В следующих разделах мы рассмотрим шаги по подготовке данных обучения, созданию сценария обучения и запуску задания обучения SageMaker. Весь код, представленный в этом посте, доступен на GitHub.
Подготовьте данные для обучения
Мы используем часть Набор данных DeepLoc-2, который содержит несколько тысяч белков SwissProt с экспериментально определенным расположением. Мы фильтруем высококачественные последовательности между 100–512 аминокислотами:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(("Unnamed: 0", "Partition"), axis=1)
df("Membrane") = df("Membrane").astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df(df("Sequence").apply(lambda x: len(x)).between(100, 512))
# Remove unnecessary features
df = df(("Sequence", "Kingdom", "Membrane"))
Затем мы токенизируем последовательности и разделяем их на обучающий и оценочный наборы:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples("Sequence")
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding("labels") = examples("Membrane")
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset("train").column_names,
)
encoded_dataset.set_format("torch")
Наконец, мы загружаем обработанные данные обучения и оценки в Amazon Simple Storage Service (Amazon S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset("train").save_to_disk(train_s3_uri)
encoded_dataset("test").save_to_disk(test_s3_uri)Создайте сценарий обучения
Режим сценариев SageMaker позволяет запускать собственный обучающий код в оптимизированных контейнерах платформы машинного обучения (ML), управляемых AWS. Для этого примера мы адаптируем существующий скрипт для классификации текста из «Обнимающего лица». Это позволяет нам попробовать несколько методов повышения эффективности нашей учебной работы.
Метод 1: Тренировочный класс с взвешиванием
Как и во многих наборах биологических данных, данные DeepLoc распределены неравномерно, то есть не существует одинакового количества мембранных и немембранных белков. Мы могли бы выполнить повторную выборку наших данных и исключить записи из класса большинства. Однако это уменьшит общий объем обучающих данных и потенциально ухудшит нашу точность. Вместо этого мы вычисляем веса классов во время обучающей работы и используем их для корректировки потерь.
В нашем сценарии обучения мы создаем подклассы Trainer класс из transformers с WeightedTrainer класс, который учитывает веса классов при расчете потерь перекрестной энтропии. Это помогает предотвратить предвзятость в нашей модели:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossМетод 2: Накопление градиента
Накопление градиента — это метод обучения, который позволяет моделям имитировать обучение в пакетах большего размера. Обычно размер пакета (количество выборок, используемых для расчета градиента за один шаг обучения) ограничен объемом памяти графического процессора. При накоплении градиента модель сначала рассчитывает градиенты для небольших партий. Затем вместо немедленного обновления весов модели градиенты накапливаются в нескольких небольших пакетах. Когда накопленные градиенты равны целевому большему размеру партии, выполняется этап оптимизации для обновления модели. Это позволяет моделям эффективно тренироваться с более крупными пакетами, не превышая предел памяти графического процессора.
Однако для меньших пакетных проходов вперед и назад необходимы дополнительные вычисления. Увеличение размеров пакетов за счет накопления градиента может замедлить обучение, особенно если используется слишком много шагов накопления. Цель состоит в том, чтобы максимизировать использование графического процессора, но избежать чрезмерного замедления из-за слишком большого количества дополнительных шагов вычисления градиента.
Метод 3: контрольная точка градиента
Градиентная контрольная точка — это метод, который уменьшает объем памяти, необходимой во время обучения, сохраняя при этом разумное время вычислений. Большие нейронные сети занимают много памяти, поскольку им приходится хранить все промежуточные значения прямого прохода, чтобы вычислить градиенты во время обратного прохода. Это может вызвать проблемы с памятью. Одно из решений — не сохранять эти промежуточные значения, но тогда их придется пересчитывать во время обратного прохода, что занимает много времени.
Градиентная контрольная точка обеспечивает сбалансированный подход. Он сохраняет только некоторые промежуточные значения, называемые контрольно-пропускные пунктыи пересчитывает остальные по мере необходимости. Следовательно, он использует меньше памяти, чем все хранение, но и меньше вычислений, чем все пересчеты. Стратегически выбирая, какие активации следует проверять, градиентная контрольная точка позволяет обучать большие нейронные сети с управляемым использованием памяти и временем вычислений. Этот важный метод позволяет обучать очень большие модели, которые в противном случае столкнулись бы с ограничениями памяти.
В нашем обучающем скрипте мы включаем активацию градиента и контрольную точку, добавляя необходимые параметры в TrainingArguments объект:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Метод 4: Адаптация LLM низкого ранга
Большие языковые модели, такие как ESM-2, могут содержать миллиарды параметров, обучение и запуск которых требуют больших затрат. Исследователи разработал метод обучения под названием «Низкоранговая адаптация» (LoRA), чтобы сделать тонкую настройку этих огромных моделей более эффективной.
Ключевая идея LoRA заключается в том, что при точной настройке модели под конкретную задачу вам не нужно обновлять все исходные параметры. Вместо этого LoRA добавляет в модель новые матрицы меньшего размера, которые преобразуют входные и выходные данные. Во время точной настройки обновляются только эти матрицы меньшего размера, что происходит намного быстрее и требует меньше памяти. Исходные параметры модели остаются замороженными.
После точной настройки с помощью LoRA вы можете объединить небольшие адаптированные матрицы обратно в исходную модель. Или вы можете оставить их отдельно, если хотите быстро настроить модель для других задач, не забывая о предыдущих. В целом, LoRA позволяет LLM эффективно адаптироваться к новым задачам за небольшую часть обычных затрат.
В нашем сценарии обучения мы настраиваем LoRA, используя PEFT библиотека из Hugging Face:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=(
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
)
)
model = get_peft_model(model, peft_config)Отправьте задание на обучение SageMaker
После определения сценария обучения вы можете настроить и отправить задание обучения SageMaker. Сначала укажите гиперпараметры:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}Затем определите, какие показатели следует фиксировать из журналов обучения:
metric_definitions = (
{"Name": "epoch", "Regex": "'epoch': ((0-9.)*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ((0-9.e-)*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ((0-9.e-)*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ((0-9.e-)*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ((0-9.e-)*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ((0-9.e-)*)"},
)Наконец, определите оценщик Hugging Face и отправьте его для обучения на экземпляре типа ml.g5.2xlarge. Это экономичный тип инстанса, широко доступный во многих регионах AWS:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=({"Key": "project", "Value": "esm-fine-tuning"}),
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)В следующей таблице сравниваются различные методы обучения, которые мы обсуждали, и их влияние на время выполнения, точность и требования к памяти графического процессора для нашей работы.
| Конфигурация | Оплачиваемое время (мин) | Точность оценки | Максимальное использование памяти графического процессора (ГБ) |
| Базовая модель | 28 | 0,91 | 22,6 |
| База + GA | 21 | 0,90 | 17,8 |
| База + GC | 29 | 0,91 | 10.2 |
| База + ЛоРА | 23 | 0,90 | 18,6 |
Все методы позволили получить модели с высокой точностью оценки. Использование LoRA и активации градиента сократило время выполнения (и стоимость) на 18% и 25% соответственно. Использование контрольных точек градиента снизило максимальное использование памяти графического процессора на 55%. В зависимости от ваших ограничений (стоимость, время, аппаратное обеспечение) один из этих подходов может иметь больше смысла, чем другой.
Каждый из этих методов хорошо работает сам по себе, но что произойдет, если мы будем использовать их в сочетании? В следующей таблице суммированы результаты.
| Конфигурация | Оплачиваемое время (мин) | Точность оценки | Максимальное использование памяти графического процессора (ГБ) |
| Все методы | 12 | 0,80 | 3.3 |
В этом случае мы видим снижение точности на 12%. Однако мы сократили время выполнения на 57 % и использование памяти графического процессора на 85 %! Это значительное снижение позволяет нам обучаться на широком спектре экономичных типов инстансов.
Очистить
Если вы следите за своими действиями в своей учетной записи AWS, удалите все созданные вами конечные точки вывода в реальном времени и данные, чтобы избежать дальнейших расходов.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()Заключение
В этом посте мы продемонстрировали, как эффективно настроить модели белкового языка, такие как ESM-2, для решения научно значимой задачи. Дополнительную информацию об использовании библиотек Transformers и PEFT для обучения pLMS можно найти в публикациях. Глубокое обучение с белками и ESMBind (ESMB): низкоранговая адаптация ESM-2 для прогнозирования сайта связывания белков в блоге Hugging Face. Вы также можете найти больше примеров использования машинного обучения для прогнозирования свойств белка в Потрясающий анализ белков на AWS Репозиторий GitHub.
об авторе
 Брайан Лоял — старший архитектор решений AI/ML в отделе глобального здравоохранения и биологических наук в Amazon Web Services. Он имеет более чем 17-летний опыт работы в области биотехнологий и машинного обучения и с энтузиазмом помогает клиентам решать геномные и протеомные проблемы. В свободное время он любит готовить и есть с друзьями и семьей.
Брайан Лоял — старший архитектор решений AI/ML в отделе глобального здравоохранения и биологических наук в Amazon Web Services. Он имеет более чем 17-летний опыт работы в области биотехнологий и машинного обучения и с энтузиазмом помогает клиентам решать геномные и протеомные проблемы. В свободное время он любит готовить и есть с друзьями и семьей.