Метод AlphaFold
Многие новые инновации в области машинного обучения способствуют текущему уровню точности AlphaFold. Мы даем общий обзор системы ниже; техническое описание сетевой архитектуры см. в нашем AlphaFold методическая статья и особенно его обширную дополнительную информацию.
Сеть AlphaFold состоит из двух основных этапов. Стадия 1 принимает в качестве входных данных аминокислотную последовательность и множественное выравнивание последовательностей (MSA). Его цель — изучить богатое «парное представление», которое дает информацию о том, какие пары остатков близки в трехмерном пространстве.
Этап 2 использует это представление для непосредственного получения атомных координат, рассматривая каждый остаток как отдельный объект, предсказывая вращение и перемещение, необходимые для размещения каждого остатка, и, в конечном итоге, собирая структурированную цепочку. Дизайн сети опирается на наши интуитивные представления о физике и геометрии белков, например, в виде применяемых обновлений и выбора потерь.
Интересно, что мы можем создать трехмерную структуру на основе представления на промежуточных уровнях сети. Полученные видео «траектории» показывают, как убеждение AlphaFold о правильной структуре развивается во время логического вывода, слой за слоем. Обычно гипотеза появляется после первых нескольких слоев, за которыми следует длительный процесс уточнения, хотя для некоторых целей требуется полная глубина сети, чтобы получить хороший прогноз.
Точность и уверенность
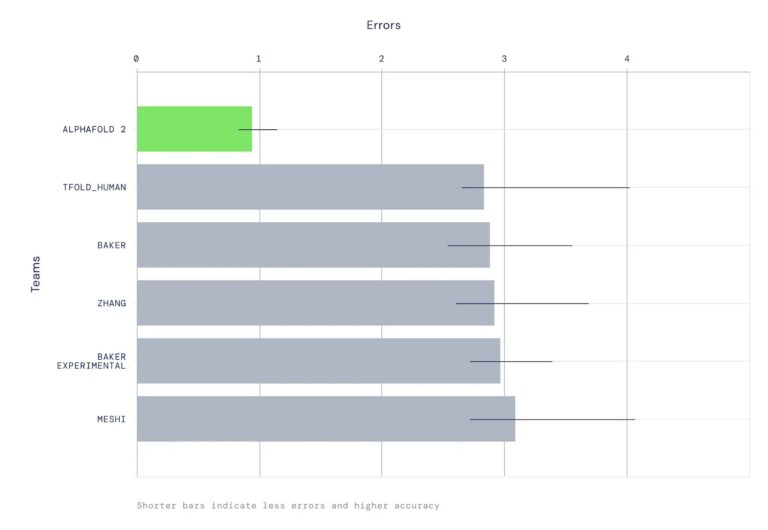
AlphaFold был строго оценен в CASP14 эксперимент, в котором участники вслепую предсказывают белковые структуры, которые были решены, но еще не обнародованы. Метод достиг высокой точности в большинстве случаев со средним 95% RMSD-Cα для экспериментальной структуры менее 1 Å. В наших статьях мы дополнительно оцениваем модель на гораздо большем наборе недавних записей PDB. Среди результатов — высокая производительность на больших белках и хорошая точность боковой цепи, где хорошо предсказана основная цепь.

Важным фактором полезности структурных прогнозов является качество соответствующих мер достоверности. Может ли модель определить части своего прогноза, которые могут быть надежными? Для ответа на этот вопрос мы разработали две меры доверия поверх сети AlphaFold.
Первый – это pLDDT (предсказанный lDDT-Cα), мера локальной достоверности для каждого остатка по шкале от 0 до 100. pLDDT может сильно различаться по цепи, что позволяет модели выражать высокую достоверность в отношении структурированных доменов, но низкую достоверность в отношении линкеров между ними, например. В нашем бумагамы представляем доказательства того, что некоторые регионы с низким уровнем pLDDT могут быть неструктурированными изолированно; либо внутренне неупорядоченные, либо структурированные только в контексте более крупного комплекса. Области с pLDDT < 50 не следует интерпретировать, кроме как как возможный прогноз расстройства.
Вторая метрика — PAE (прогнозируемая ошибка выравнивания), которая сообщает об ожидаемой ошибке позиционирования AlphaFold в остатке x, когда предсказанная и истинная структуры выровнены в остатке y. Это полезно для оценки достоверности глобальных функций, особенно упаковки доменов. Для остатков x и y, взятых из двух разных доменов, постоянно низкий PAE в (x, y) свидетельствует о том, что AlphaFold уверен в относительном расположении доменов. Постоянно высокий PAE в (x, y) предполагает, что относительное положение доменов не следует интерпретировать. Общий подход, используемый для получения PAE, может быть адаптирован для прогнозирования различных показателей на основе суперпозиции, включая ТМ-оценка и ГДТ.
.jpg)
Подчеркнем, что модели AlphaFold в конечном счете являются прогнозами: хотя они часто очень точны, иногда они бывают ошибочными. Предсказанные координаты атомов следует интерпретировать осторожно и в контексте этих доверительных мер.
Открытый исходный код
Наряду с нашим метод документмы сделали исходный код AlphaFold доступным на Гитхаб. Это включает в себя доступ к обученной модели и скрипту для прогнозирования новых входных последовательностей. Мы считаем, что это важный шаг, который позволит сообществу использовать и развивать нашу работу. Самый простой способ сложить один новый белок с помощью AlphaFold — использовать нашу Блокнот Colab.
Открытый исходный код представляет собой обновленную версию нашей системы CASP14, основанную на JAX-фреймворк, и достигается столь же высокая точность. Он также включает в себя некоторые недавние улучшения производительности. Скорость AlphaFold всегда сильно зависела от длины входной последовательности: для обработки коротких белков требуются минуты, а для обработки очень длинных белков — часы. После сборки MSA версия с открытым исходным кодом теперь может предсказывать структуру белка из 400 остатков чуть более чем за минуту работы графического процессора на V100.
Шкала протеома и база данных AlphaFold
Быстрое время вывода AlphaFold позволяет применять метод в масштабе всего протеома. В нашем бумага, мы обсуждаем прогнозы AlphaFold для человеческого протеома. Однако с тех пор мы создали прогнозы для эталонных протеомов ряда модельные организмы, патогены и экономически значимые виды, и крупномасштабное предсказание теперь является обычным делом. Интересно, что мы наблюдаем разницу в распределении pLDDT между видами, с более высокой достоверностью для бактерий и архей и меньшей достоверностью для эукариот, что, как мы предполагаем, может быть связано с преобладанием беспорядка в этих протеомах.
Ни одна исследовательская группа не может полностью изучить такой большой набор данных, поэтому мы сотрудничали с ЭМБЛ-ЭБИ делать прогнозы в свободном доступе через БД AlphaFold. Каждый прогноз можно просмотреть вместе с метриками достоверности, описанными выше. Для каждого вида также предоставляется массовая загрузка, и все данные подпадают под действие лицензии CC-BY-4.0 (что делает их бесплатными как для академического, так и для коммерческого использования). Мы чрезвычайно благодарны EMBL-EBI за их работу с нами по разработке этого нового ресурса. В ближайшие месяцы мы планируем расширить набор данных, чтобы охватить более 100 миллионов белков в UniRef90.
.jpg)
.jpg)
В базе данных AlphaFold мы решили делиться прогнозами полных белковых цепей длиной до 2700 аминокислот, а не обрезать отдельные домены. Смысл в том, что это позволяет избежать пропуска структурированных областей, которые еще не аннотированы. Он также предоставляет контекст из полной аминокислотной последовательности и позволяет модели попытаться предсказать упаковку доменов. Внутридоменная точность AlphaFold была более тщательно оценена в CASP14 и, как ожидается, будет выше, чем его междоменная точность. Тем не менее, AlphaFold был лучшим методом междоменной оценки, и мы ожидаем, что в некоторых случаях он будет давать информативный прогноз. Мы рекомендуем пользователям просматривать график PAE, чтобы определить, имеет ли значение размещение домена.
Будущая работа
Мы с нетерпением ждем будущего вычислительной структурной биологии. Остается еще много важных тем для решения: предсказание структуры комплексов, включение небелковых компонентов, а также учет динамики и реакции на точечные мутации. Разработка сетевых архитектур, таких как AlphaFold, которые превосходно справляются с задачей понимания структуры белка, является причиной для оптимизма в отношении того, что мы можем добиться прогресса в решении связанных проблем.
Мы рассматриваем AlphaFold как дополнительную технологию к экспериментальной структурной биологии. Это, пожалуй, лучше всего иллюстрируется его ролью в решении экспериментальных структур посредством молекулярной замены и стыковки с объемами крио-ЭМ. Оба приложения могут ускорить существующие исследования, экономя месяцы усилий. С точки зрения биоинформатики скорость AlphaFold позволяет генерировать предсказанные структуры в больших масштабах. Это может открыть новые направления исследований, поддерживая структурные исследования содержимого больших баз данных последовательностей.
В конечном счете, мы надеемся, что AlphaFold окажется полезным инструментом для освещения белкового пространства, и с нетерпением ждем возможности увидеть, как он будет применяться в ближайшие месяцы и годы.
Мы хотели бы услышать ваши отзывы и понять, как AlphaFold и база данных AlphaFold были полезны в ваших исследованиях. Поделитесь своими историями на alphafold@deepmind.com.