Этот пост написан в соавторстве с Мингом (Мелвином) Цином, Дэвидом Берикатом и Брэдом Дженеро из NVIDIA.
Исследователям и разработчикам искусственного интеллекта в области медицинских изображений необходима масштабируемая корпоративная среда для создания, развертывания и интеграции своих приложений искусственного интеллекта. AWS и NVIDIA объединились, чтобы воплотить эту идею в жизнь. AWS, NVIDIA и другие партнеры создают приложения и решения, чтобы сделать здравоохранение более доступным, экономичным и эффективным за счет ускорения подключения к облаку для корпоративных изображений. МОНАИ Развертывание является одним из ключевых модулей внутри МОНАИ (Медицинская открытая сеть для искусственного интеллекта), разработанная консорциумом академических и отраслевых лидеров, включая NVIDIA. AWS HealthImaging (AHI) — это масштабируемый, производительный и экономичный магазин медицинских изображений, отвечающий требованиям HIPAA. Мы разработали соединитель MONAI Deploy для AHI для интеграции приложений искусственного интеллекта для медицинских изображений с задержкой получения изображений в доли секунды в масштабе, основанном на облачных API. Модели и приложения MONAI AI можно разместить на Amazon SageMaker, который представляет собой полностью управляемый сервис для развертывания моделей машинного обучения (ML) в большом масштабе. SageMaker берет на себя настройку и управление экземплярами для вывода и предоставляет встроенные метрики и журналы для конечных точек, которые вы можете использовать для мониторинга и получения оповещений. Он также предлагает различные экземпляры графических процессоров NVIDIA для вывода ML, а также несколько вариантов развертывания моделей с автоматическим масштабированием, включая вывод в реальном времени, бессерверный вывод, асинхронный вывод и пакетное преобразование.
В этом посте мы демонстрируем, как развернуть пакет приложений MONAI (MAP) с соединителем для AWS HealthImaging, используя многомодельную конечную точку SageMaker для вывода в реальном времени и асинхронного вывода. Эти два варианта охватывают большинство случаев использования конвейера вывода медицинских изображений, работающих в режиме, близком к реальному времени.
Обзор решения
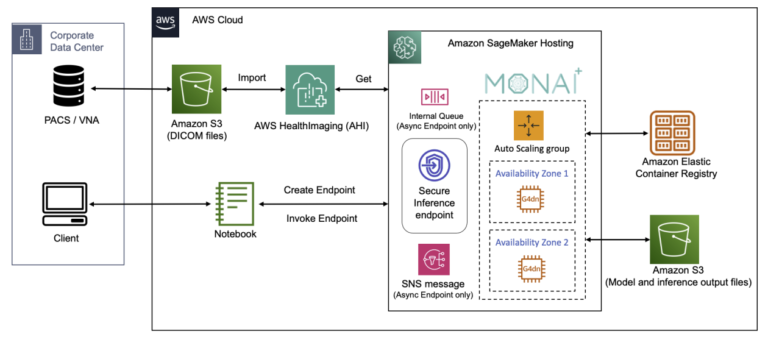
На следующей диаграмме показана архитектура решения.
Предварительные условия
Выполните следующие необходимые шаги:
- Используйте учетную запись AWS в одном из следующих регионов, где доступен AWS HealthImaging: Северная Вирджиния (
us-east-1), Орегон (us-west-2), Ирландия (eu-west-1) и Сидней (ap-southeast-2). - Создайте домен Amazon SageMaker Studio и профиль пользователя с разрешением AWS Identity and Access Management (IAM) для доступа к AWS HealthImaging.
- Включите расширение JupyterLab v3 и установите расширение Imjoy-jupyter, если вы хотите интерактивно визуализировать медицинские изображения в блокноте SageMaker с помощью itkwidgets.
Коннектор MAP к AWS HealthImaging
AWS HealthImaging импортирует файлы DICOM P10 и преобразует их в наборы изображений, которые представляют собой оптимизированное представление серии DICOM. AHI обеспечивает доступ через API к метаданным ImageSet и ImageFrames. Метаданные содержат все атрибуты DICOM в документе JSON. ImageFrames возвращаются в кодировке Высокопроизводительный JPEG2000 (HTJ2K) формат без потерь, который можно декодировать очень быстро. Наборы изображений можно получить с помощью интерфейса командной строки AWS (AWS CLI) или пакетов AWS SDK.
MONAI — это платформа искусственного интеллекта для медицинской визуализации, которая воплощает научные открытия и приложения искусственного интеллекта в клиническое применение. MONAI Deploy — это конвейер обработки, который обеспечивает сквозной рабочий процесс, включая упаковку, тестирование, развертывание и запуск приложений искусственного интеллекта для медицинских изображений в клиническом производстве. Он включает в себя МОНАИ Развертывание App SDK, MONAI Deploy Express, Менеджер рабочих процессови Информатический шлюз. SDK MONAI Deploy App предоставляет готовые к использованию алгоритмы и платформу для ускорения создания приложений искусственного интеллекта для медицинских изображений, а также служебные инструменты для упаковки приложения в контейнер MAP. Встроенные основанные на стандартах функции в SDK приложения позволяют MAP плавно интегрироваться в ИТ-сети здравоохранения, что требует использования таких стандартов, как DICOM, HL7 и FHIR, а также в центрах обработки данных и облачных средах. MAP могут использовать как предопределенные, так и настраиваемые операторы для загрузки изображений DICOM, выбора серии, вывода модели и постобработки.
Мы разработали Модуль Python с помощью AWS HealthImaging Python SDK Boto3. Вы можете установить его и использовать вспомогательную функцию для получения экземпляров пары сервис-объект (SOP) DICOM следующим образом:
!pip install -q AHItoDICOMInterface
from AHItoDICOMInterface.AHItoDICOM import AHItoDICOM
helper = AHItoDICOM()
instances = helper.DICOMizeImageSet(datastore_id=datastoreId , image_set_id=next(iter(imageSetIds)))Выходные экземпляры СОП можно визуализировать с помощью интерактивного средства просмотра медицинских 3D-изображений itkwidgets следующим образом. блокнот. АХИТОДИКОМ класс использует преимущества нескольких процессов для параллельного получения кадров пикселей из AWS HealthImaging и декодировать тот Двоичные объекты HTJ2K используя Библиотека Python OpenJPEG. ImageSetIds берутся из выходных файлов данного задания импорта AWS HealthImaging. Учитывая DatastoreId и импорт JobId, вы можете получить ImageSetId, который эквивалентен UID экземпляра серии DICOM, следующим образом:
imageSetIds = {}
try:
response = s3.head_object(Bucket=OutputBucketName, Key=f"output/{res_createstore('datastoreId')}-DicomImport-{res_startimportjob('jobId')}/job-output-manifest.json")
if response('ResponseMetadata')('HTTPStatusCode') == 200:
data = s3.get_object(Bucket=OutputBucketName, Key=f"output/{res_createstore('datastoreId')}-DicomImport-{res_startimportjob('jobId')}/SUCCESS/success.ndjson")
contents = data('Body').read().decode("utf-8")
for l in contents.splitlines():
isid = json.loads(l)('importResponse')('imageSetId')
if isid in imageSetIds:
imageSetIds(isid)+=1
else:
imageSetIds(isid)=1
except ClientError:
passС помощью ImageSetId вы можете получать метаданные заголовка DICOM и пиксели изображения отдельно, используя собственные функции API AWS HealthImaging. Агрегаты экспортеров DICOM заголовки DICOM и пиксели изображения в Пидиком набор данных, который может быть обработан Оператор загрузчика данных MAP DICOM. Используя функцию DICOMizeImageSet(), мы создали коннектор для загрузки данных изображения из AWS HealthImaging на основе MAP. Оператор загрузчика данных DICOM:
class AHIDataLoaderOperator(Operator):
def __init__(self, ahi_client, must_load: bool = True, *args, **kwargs):
self.ahi_client = ahi_client
…
def _load_data(self, input_obj: string):
study_dict = {}
series_dict = {}
sop_instances = self.ahi_client.DICOMizeImageSet(input_obj('datastoreId'), input_obj('imageSetId'))В предыдущем коде ahi_client является экземпляром класса экспортера DICOM AHItoDICOM с иллюстрированными функциями поиска данных. Мы включили этот новый оператор загрузки данных в AI-приложение для 3D-сегментации селезенки, созданное с помощью MONAI Deploy App SDK. Сначала вы можете изучить, как создать и запустить это приложение. в экземпляре локального блокнотаа затем разверните это приложение MAP в конечных точках управляемого вывода SageMaker.
Асинхронный вывод SageMaker
Конечная точка асинхронного вывода SageMaker используется для запросов с большими размерами полезных данных (до 1 ГБ), длительным временем обработки (до 15 минут) и требованиями к задержке, близкой к реальному времени. Когда нет запросов для обработки, этот вариант развертывания может уменьшить количество экземпляров до нуля для экономии средств, что идеально подходит для рабочих нагрузок машинного обучения в области медицинских изображений. Следуйте инструкциям в образец блокнота для создания и вызова конечной точки асинхронного вывода SageMaker. Чтобы создать конечную точку асинхронного вывода, вам необходимо сначала создать модель SageMaker и конфигурацию конечной точки. Чтобы создать модель SageMaker, вам необходимо загрузить пакет model.tar.gz с определенная структура каталогов в контейнер Docker. Пакет model.tar.gz включает предварительно обученный файл model.ts сегментации селезенки и настроенный файл inference.py. Мы использовали готовый контейнер с версиями платформы Python 3.8 и PyTorch 1.12.1 для загрузки модели и выполнения прогнозов.
В индивидуальном inference.py мы создаем экземпляр вспомогательного класса AHItoDICOM из AHItoDICOMInterface и используем его для создания экземпляра MAP в model_fn() функцию, и мы запускаем приложение MAP при каждом запросе вывода в predict_fn() функция:
from app import AISpleenSegApp
from AHItoDICOMInterface.AHItoDICOM import AHItoDICOM
helper = AHItoDICOM()
def model_fn(model_dir, context):
…
monai_app_instance = AISpleenSegApp(helper, do_run=False,path="/home/model-server")
def predict_fn(input_data, model):
with open('/home/model-server/inputImageSets.json', 'w') as f:
f.write(json.dumps(input_data))
output_folder = "/home/model-server/output"
if not os.path.exists(output_folder):
os.makedirs(output_folder)
model.run(input="/home/model-server/inputImageSets.json", output=output_folder, workdir="/home/model-server", model="/opt/ml/model/model.ts")Чтобы вызвать асинхронную конечную точку, вам необходимо загрузить входные данные запроса в Amazon Simple Storage Service (Amazon S3), который представляет собой файл JSON, в котором указаны идентификатор хранилища данных AWS HealthImaging и идентификатор ImageSet для выполнения вывода:
sess = sagemaker.Session()
InputLocation = sess.upload_data('inputImageSets.json', bucket=sess.default_bucket(), key_prefix=prefix, extra_args={"ContentType": "application/json"})
response = runtime_sm_client.invoke_endpoint_async(EndpointName=endpoint_name, InputLocation=InputLocation, ContentType="application/json", Accept="application/json")
output_location = response("OutputLocation")Вывод также можно найти в Amazon S3.
Многомодельный вывод SageMaker в реальном времени
Конечные точки вывода SageMaker в реальном времени соответствуют интерактивным требованиям с малой задержкой. Этот вариант позволяет разместить несколько моделей в одном контейнере за одной конечной точкой, что является масштабируемым и экономичным решением для развертывания нескольких моделей машинного обучения. Конечная точка SageMaker с несколькими моделями использует сервер вывода NVIDIA Triton с графическим процессором для выполнения нескольких выводов модели глубокого обучения.
В этом разделе мы рассмотрим, как создать и вызвать многомодельную конечную точку, адаптируя ваш собственный контейнер вывода, как показано ниже. образец блокнота. Различные модели могут обслуживаться в общем контейнере на одном и том же парке ресурсов. Конечные точки с несколькими моделями сокращают затраты на развертывание и позволяют масштабировать модели на основе моделей трафика, поступающих к конечной точке. Мы использовали инструменты разработчика AWS, включая Amazon CodeCommit, Amazon CodeBuild и Amazon CodePipeline, чтобы создать индивидуальный контейнер для вывода моделей SageMaker. Мы подготовили model_handler.py чтобы использовать собственный контейнер вместо файла inference.py в предыдущем примере, и реализовали функции Initialize(), preprocess() и inference():
from app import AISpleenSegApp
from AHItoDICOMInterface.AHItoDICOM import AHItoDICOM
class ModelHandler(object):
def __init__(self):
self.initialized = False
self.shapes = None
def initialize(self, context):
self.initialized = True
properties = context.system_properties
model_dir = properties.get("model_dir")
gpu_id = properties.get("gpu_id")
helper = AHItoDICOM()
self.monai_app_instance = AISpleenSegApp(helper, do_run=False, path="/home/model-server/")
def preprocess(self, request):
inputStr = request(0).get("body").decode('UTF8')
datastoreId = json.loads(inputStr)('inputs')(0)('datastoreId')
imageSetId = json.loads(inputStr)('inputs')(0)('imageSetId')
with open('/tmp/inputImageSets.json', 'w') as f:
f.write(json.dumps({"datastoreId": datastoreId, "imageSetId": imageSetId}))
return '/tmp/inputImageSets.json'
def inference(self, model_input):
self.monai_app_instance.run(input=model_input, output="/home/model-server/output/", workdir="/home/model-server/", model=os.environ("model_dir")+"/model.ts")После того как контейнер будет создан и отправлен в реестр Amazon Elastic Container Registry (Amazon ECR), вы сможете создать с его помощью модель SageMaker, а также различные пакеты моделей (файлы tar.gz) по заданному пути Amazon S3:
model_name = "DEMO-MONAIDeployModel" + strftime("%Y-%m-%d-%H-%M-%S", gmtime())
model_url = "s3://{}/{}/".format(bucket, prefix)
container = "{}.dkr.ecr.{}.amazonaws.com/{}:dev".format( account_id, region, prefix )
container = {"Image": container, "ModelDataUrl": model_url, "Mode": "MultiModel"}
create_model_response = sm_client.create_model(ModelName=model_name, ExecutionRoleArn=role, PrimaryContainer=container)Примечательно, что model_url здесь указывается только путь к папке с файлами tar.gz, и вы указываете, какой пакет модели использовать для вывода при вызове конечной точки, как показано в следующем коде:
Payload = {"inputs": ( {"datastoreId": datastoreId, "imageSetId": next(iter(imageSetIds))} )}
response = runtime_sm_client.invoke_endpoint(EndpointName=endpoint_name, ContentType="application/json", Accept="application/json", TargetModel="model.tar.gz", Body=json.dumps(Payload))Мы можем добавить больше моделей к существующей конечной точке многомодельного вывода без необходимости обновлять конечную точку или создавать новую.
Очистить
Не забудьте завершить Удалить ресурсы хостинга вступить в лаборатория-3 и лаборатория-4 блокноты, чтобы удалить конечные точки вывода SageMaker. Вам также следует отключить экземпляр блокнота SageMaker, чтобы сэкономить средства. Наконец, вы можете либо вызвать функцию API AWS HealthImaging, либо использовать консоль AWS HealthImaging для удаления наборов изображений и хранилища данных, созданных ранее:
for s in imageSetIds.keys():
medicalimaging.deleteImageSet(datastoreId, s)
medicalimaging.deleteDatastore(datastoreId)Заключение
В этом посте мы показали, как создать соединитель MAP для AWS HealthImaging, который можно повторно использовать в приложениях, созданных с помощью MONAI Deploy App SDK, для интеграции и ускорения получения данных изображений из облачного хранилища DICOM для рабочих нагрузок искусственного интеллекта в области медицинских изображений. . MONAI Deploy SDK можно использовать для поддержки работы больниц. Мы также продемонстрировали два варианта хостинга для масштабного развертывания приложений MAP AI на SageMaker.
Просмотрите примеры тетрадей в Репозиторий GitHub чтобы узнать больше о том, как развертывать приложения MONAI в SageMaker с медицинскими изображениями, хранящимися в AWS HealthImaging. Чтобы узнать, чем AWS может вам помочь, обратитесь к Представитель AWS.
Дополнительные ресурсы см. в следующих разделах:
Об авторах
 Мин (Мелвин) Цинь — независимый участник группы здравоохранения NVIDIA, занимающейся разработкой среды и платформы приложений для вывода ИИ, позволяющих использовать ИИ в рабочих процессах медицинской визуализации. Прежде чем присоединиться к NVIDIA в 2018 году в качестве одного из основателей компании Clara, Минг в течение 15 лет занимался разработкой радиологических систем PACS и Workflow SaaS в качестве ведущего инженера/архитектора в компании Stentor Inc., которую позже приобрела компания Philips Healthcare для формирования подразделения Enterprise Imaging.
Мин (Мелвин) Цинь — независимый участник группы здравоохранения NVIDIA, занимающейся разработкой среды и платформы приложений для вывода ИИ, позволяющих использовать ИИ в рабочих процессах медицинской визуализации. Прежде чем присоединиться к NVIDIA в 2018 году в качестве одного из основателей компании Clara, Минг в течение 15 лет занимался разработкой радиологических систем PACS и Workflow SaaS в качестве ведущего инженера/архитектора в компании Stentor Inc., которую позже приобрела компания Philips Healthcare для формирования подразделения Enterprise Imaging.
 Дэвид Берикат — менеджер по продуктам в сфере здравоохранения в компании NVIDIA, где он возглавляет рабочую группу Project MONAI Deploy, призванную перенести ИИ из исследований в клиническое развертывание. Его страсть — ускорять инновации в области здравоохранения во всем мире, превращая их в настоящий клинический эффект. Ранее Дэвид работал в Red Hat, внедряя принципы открытого исходного кода на стыке искусственного интеллекта, облака, периферийных вычислений и Интернета вещей. Моментами, которыми он больше всего гордится, являются походы к базовому лагерю Эвереста и игра в футбол на протяжении более 20 лет.
Дэвид Берикат — менеджер по продуктам в сфере здравоохранения в компании NVIDIA, где он возглавляет рабочую группу Project MONAI Deploy, призванную перенести ИИ из исследований в клиническое развертывание. Его страсть — ускорять инновации в области здравоохранения во всем мире, превращая их в настоящий клинический эффект. Ранее Дэвид работал в Red Hat, внедряя принципы открытого исходного кода на стыке искусственного интеллекта, облака, периферийных вычислений и Интернета вещей. Моментами, которыми он больше всего гордится, являются походы к базовому лагерю Эвереста и игра в футбол на протяжении более 20 лет.
 Брэд Женеро является глобальным руководителем отдела медицинских альянсов в NVIDIA, где он отвечает за отношения с разработчиками, специализирующимися на медицинской визуализации для ускорения искусственного интеллекта и глубокого обучения, визуализации, виртуализации и аналитических решений. Брэд пропагандирует повсеместное внедрение и интеграцию бесперебойных рабочих процессов в сфере здравоохранения и медицинской визуализации в повседневную клиническую практику, имея более чем 20-летний опыт работы в сфере ИТ в сфере здравоохранения.
Брэд Женеро является глобальным руководителем отдела медицинских альянсов в NVIDIA, где он отвечает за отношения с разработчиками, специализирующимися на медицинской визуализации для ускорения искусственного интеллекта и глубокого обучения, визуализации, виртуализации и аналитических решений. Брэд пропагандирует повсеместное внедрение и интеграцию бесперебойных рабочих процессов в сфере здравоохранения и медицинской визуализации в повседневную клиническую практику, имея более чем 20-летний опыт работы в сфере ИТ в сфере здравоохранения.
 Ган Фу — архитектор решений для здравоохранения в AWS. Он получил степень доктора фармацевтических наук в Университете Миссисипи и имеет более чем 10-летний опыт работы в области технологий и биомедицинских исследований. Он увлечен технологиями и тем влиянием, которое они могут оказать на здравоохранение.
Ган Фу — архитектор решений для здравоохранения в AWS. Он получил степень доктора фармацевтических наук в Университете Миссисипи и имеет более чем 10-летний опыт работы в области технологий и биомедицинских исследований. Он увлечен технологиями и тем влиянием, которое они могут оказать на здравоохранение.
 Ж. П. Леже — старший архитектор решений, поддерживающий академические медицинские центры и рабочие процессы медицинской визуализации в AWS. Он обладает более чем 20-летним опытом разработки программного обеспечения, ИТ в сфере здравоохранения и медицинской визуализации, а также обширным опытом проектирования систем для обеспечения производительности, масштабируемости и безопасности в распределенных развертываниях больших объемов данных локально, в облаке и в гибридных системах с аналитикой и искусственным интеллектом. .
Ж. П. Леже — старший архитектор решений, поддерживающий академические медицинские центры и рабочие процессы медицинской визуализации в AWS. Он обладает более чем 20-летним опытом разработки программного обеспечения, ИТ в сфере здравоохранения и медицинской визуализации, а также обширным опытом проектирования систем для обеспечения производительности, масштабируемости и безопасности в распределенных развертываниях больших объемов данных локально, в облаке и в гибридных системах с аналитикой и искусственным интеллектом. .
 Крис Хэфи — главный архитектор решений в Amazon Web Services. Он имеет более чем 25-летний опыт работы в сфере медицинской визуализации и специализируется на создании масштабируемых высокопроизводительных систем. Он является создателем популярного проекта с открытым исходным кодом CornerstoneJS, который лежит в основе популярной программы просмотра с открытым исходным кодом OHIF. Он внес свой вклад в разработку спецификации DICOMweb и продолжает работать над улучшением ее производительности для просмотра через Интернет.
Крис Хэфи — главный архитектор решений в Amazon Web Services. Он имеет более чем 25-летний опыт работы в сфере медицинской визуализации и специализируется на создании масштабируемых высокопроизводительных систем. Он является создателем популярного проекта с открытым исходным кодом CornerstoneJS, который лежит в основе популярной программы просмотра с открытым исходным кодом OHIF. Он внес свой вклад в разработку спецификации DICOMweb и продолжает работать над улучшением ее производительности для просмотра через Интернет.
