Настало время для практического руководства по медицинской визуализации. Однако на этот раз мы будем использовать не сумасшедший ИИ, а базовые алгоритмы обработки изображений. Цель состоит в том, чтобы познакомить читателя с концепциями медицинской визуализации и, в частности, компьютерной томографии (КТ).
Важно понять, как далеко можно зайти без глубокое обучение, чтобы понять, когда лучше его использовать. Новые практикующие врачи склонны игнорировать эту часть, но анализ медицинских изображений по-прежнему является обработкой трехмерных изображений.
Я также включаю части кода, чтобы облегчить понимание моего мыслительного процесса.
Сопутствующий блокнот Google Colab можно найти здесь для запуска кода, показанного в этом руководстве. А Гитхаб репозиторий также доступен. Пометьте наш репозиторий, если он вам понравился!
Чтобы глубже погрузиться в то, как ИИ используется в медицине, вы не ошибетесь с ИИ для медицины онлайн-курс, предлагаемый Coursera. Если вы хотите сосредоточиться на анализе медицинских изображений с помощью глубокого обучения, я настоятельно рекомендую начать с Курс Udemy на основе Pytorch.
Мы начнем с самых основ компьютерной томографии. Вы можете пропустить этот раздел, если вы уже знакомы с КТ.
КТ-визуализация
Физика компьютерной томографии
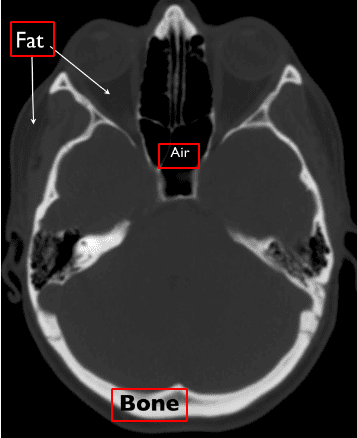
Компьютерная томография (КТ) использует рентгеновские лучи для получения трехмерной интенсивности пикселей человеческого тела. Нагретый катод испускает высокоэнергетические пучки (электроны), которые, в свою очередь, выделяют свою энергию в виде рентгеновского излучения. Рентгеновские лучи проходят через ткани человеческого тела и попадают на детектор с другой стороны. Плотная ткань (т.е. кости) будет поглощать больше радиации, чем мягкие ткани (т.е. жир). Когда рентгеновские лучи не поглощаются телом (т.е. в воздушной области легких) и достигают детектора, мы видим их черными, похожими на черную пленку. Напротив, плотные ткани изображаются белым цветом.
Таким образом, Компьютерная томография способна различать различия в плотности и создавать трехмерное изображение тела..
Вот 1-минутное видео, которое я нашел очень кратким:
Интенсивности КТ и единицы Хаунсфилда
Поглощение рентгеновского излучения измеряется в шкала Хаунсфилда. В этой шкале мы устанавливаем интенсивность воздуха на -1000 и интенсивность воды на 0. Важно понимать, что Хаузенфилд — это абсолютная шкала, в отличие от МРТ, где у нас есть относительная шкала от 0 до 255.
На изображении показаны некоторые основные ткани и соответствующие им значения интенсивности. Имейте в виду, что изображения шумные. Цифры могут немного отличаться на реальных изображениях.
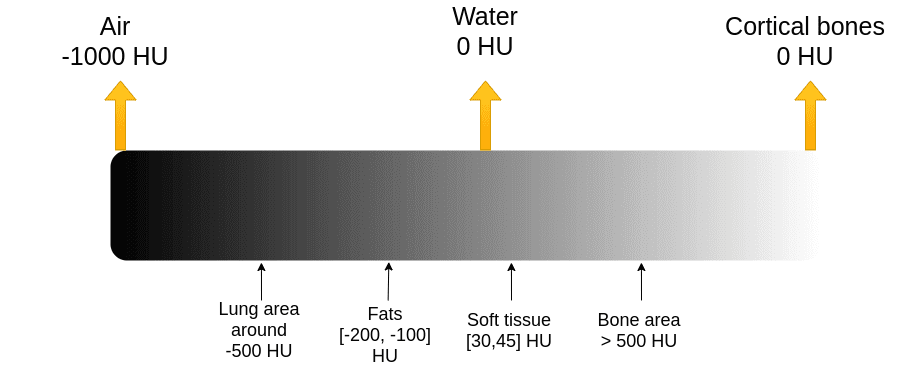
Шкала Хаунсфилда. Изображение автора.
Кости имеют высокую интенсивность. Обычно мы обрезаем изображение, чтобы иметь верхний максимальный диапазон. Например, максимальное значение может быть 1000 из практических соображений.
Проблема: библиотеки визуализации работают в масштабе (0,255). Было бы не очень разумно визуализировать всю шкалу Хаунсфилда (от -1000 до 1000+) до 256 шкал для медицинской диагностики.
Вместо этого мы ограничиваем свое внимание различными частями этого диапазона и сосредотачиваемся на подлежащих тканях.
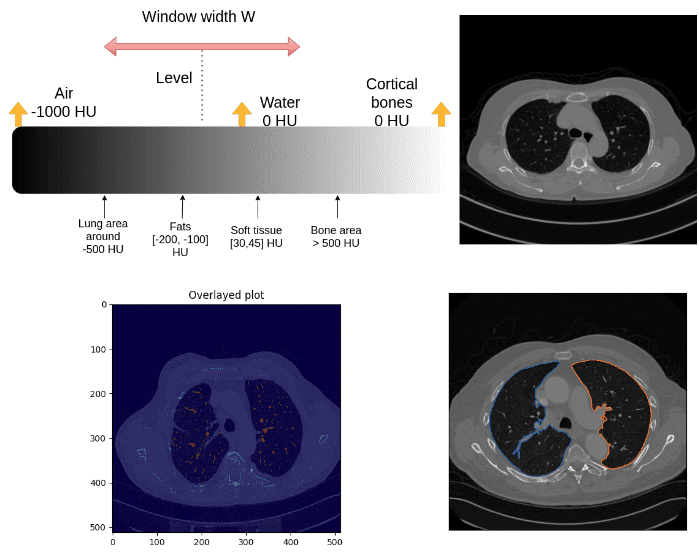
Визуализация данных КТ: уровень и окно
Соглашение о медицинском изображении для обрезки диапазона Хазенфилда заключается в выборе центральной интенсивности, называемой уровнем, и окна, как показано на рисунке:
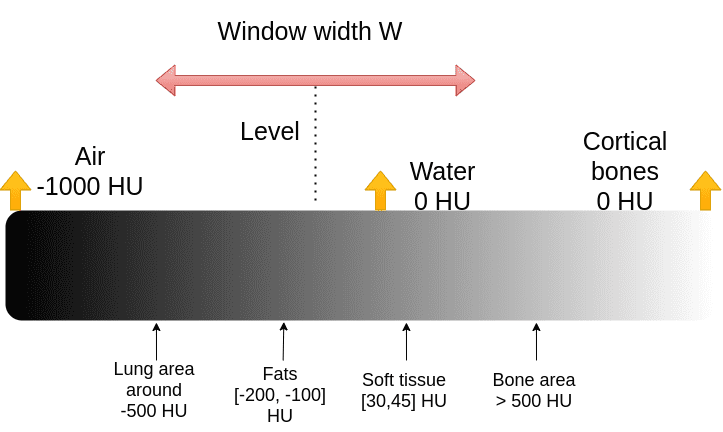
На самом деле это довольно уродливое соглашение для ученых-компьютерщиков. Нам просто нужен минимальный и максимальный диапазон:
import matplotlib.pyplot as plt
import numpy as np
def show_slice_window(slice, level, window):
"""
Function to display an image slice
Input is a numpy 2D array
"""
max = level + window/2
min = level - window/2
slice = slice.clip(min,max)
plt.figure()
plt.imshow(slice.T, cmap="gray", origin="lower")
plt.savefig('L'+str(level)+'W'+str(window))
Если вы не уверены, следующее изображение убедит вас в том, что то же изображение КТ более богато, чем обычный канал изображения:
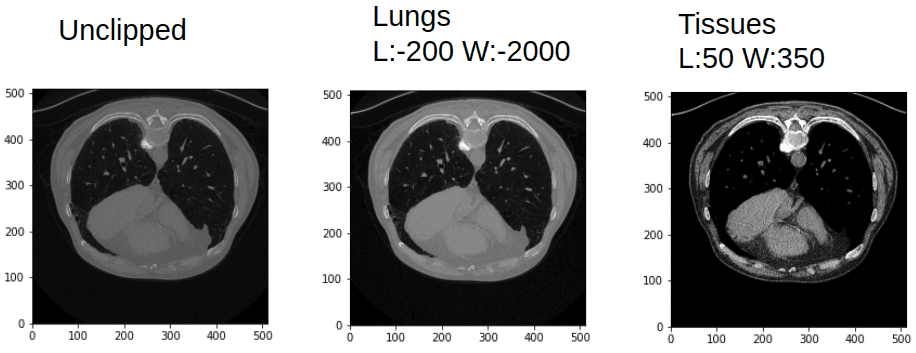
Окно и выравнивание в CT могут дать вам совершенно разные изображения. Изображение автора.
Для справки вот список диапазонов визуализации:
| Регион/ткань | Окно | Уровень |
| мозг | 80 | 40 |
| легкие | 1500 | -600 |
| печень | 150 | 30 |
| Мягкие ткани | 250 | 50 |
| кость | 1800 | 400 |
Время играть!
Сегментация легких на основе значений интенсивности
Мы не просто сегментируем легкие, но и найдем реальную площадь в . Для этого нам нужно найти реальный размер пикселей. У каждого изображения может быть свое изображение (pixdim в изящном заголовочном файле). Давайте сначала посмотрим файл заголовка:
import nibabel as nib
ct_img = nib.load(exam_path)
print(ct_img.header)
Здесь я покажу только некоторые важные поля шапки:
<class 'nibabel.nifti1.Nifti1Header'> object, endian='<'
sizeof_hdr : 348
dim : ( 2 512 512 1 1 1 1 1)
datatype : int16
bitpix : 16
pixdim : (1. 0.78515625 0.78515625 1. 1. 1. 1. 1. )
srow_x : ( -0.78515625 0. 0. 206.60742 )
srow_y : ( 0. -0.78515625 0. 405.60742 )
srow_z : ( 0. 0. 1. -304.5)
Для справки, srow_x, srow_y, srow_z — это аффинная матрица изображения. Bitpix — это количество битов, которое мы используем для представления интенсивности каждого пикселя.
Итак, давайте определим функцию, которая считывает эту информацию из заголовочного файла. На основе изящного формата каждое измерение в изящном файле имеет размер в пикселях. Что нам нужно, так это узнать 2 индекса измерения изображения и их соответствующие размеры в пикселях.
Шаг 1: Найдите размеры в пикселях, чтобы вычислить площадь в мм^2
def find_pix_dim(ct_img):
"""
Get the pixdim of the CT image.
A general solution that gets the pixdim indicated from the image dimensions. From the last 2 image dimensions, we get their pixel dimension.
Args:
ct_img: nib image
Returns: List of the 2 pixel dimensions
"""
pix_dim = ct_img.header("pixdim")
dim = ct_img.header("dim")
max_indx = np.argmax(dim)
pixdimX = pix_dim(max_indx)
dim = np.delete(dim, max_indx)
pix_dim = np.delete(pix_dim, max_indx)
max_indy = np.argmax(dim)
pixdimY = pix_dim(max_indy)
return (pixdimX, pixdimY)
Шаг 2: Бинаризация изображения с использованием порога интенсивности
Мы ожидаем, что легкие будут находиться в диапазоне единиц Хаусендфилда. (-1000,-300). Для этого нам нужно обрезать диапазон изображения до (-1000,-300) и бинаризовать значения до 0 и 1, так что мы получим что-то вроде этого:
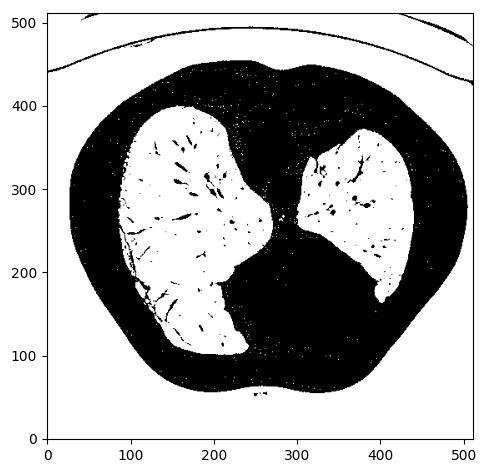
Изображение автора
Шаг 3: Определение контура
Прежде всего давайте проясним, что такое контур:
Для компьютерного зрения контур — это набор точек, описывающих линию или область. Таким образом, для каждого обнаруженного контура мы получим не полную двоичную маску, а скорее набор с набором значений x и y.
Хорошо, как мы можем изолировать нужную область? Хм.. давайте подумаем. Нас интересуют области легких, которые показаны белым цветом. Если бы мы могли найти алгоритм для определения близких наборов или любых контуров на изображении, это могло бы помочь. После недолгих поисков в Интернете я нашел метод маршевых квадратов который находит контуры с постоянными значениями на изображении из беглый просмотрназывается skimage.measure.find_contours().
После использования этой функции я визуализирую обнаруженные контуры на исходном КТ-изображении:
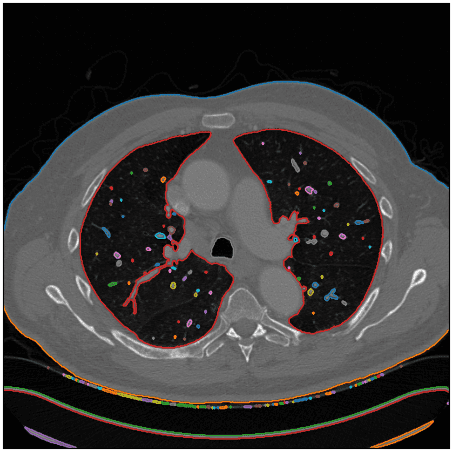
Изображение автора
Вот это функция!
def intensity_seg(ct_numpy, min=-1000, max=-300):
clipped = clip_ct(ct_numpy, min, max)
return measure.find_contours(clipped, 0.95)
Шаг 4: Найдите площадь легкого из набора возможных контуров
Обратите внимание, что я использовал другое изображение, чтобы показать пограничный случай, когда тело пациента не является замкнутым набором точек. Хорошо, не совсем то, что мы хотим, но давайте посмотрим, сможем ли мы это решить.
Поскольку нас интересуют только легкие, мы должны установить какие-то ограничения, чтобы исключить нежелательные области.
Для этого я сначала выделил из контура выпуклый многоугольник, используя острый. После того, как я предполагаю 2 ограничения:
Это может включать или не включать контур тела, в результате чего получается более 3 контуров. Когда это происходит, тело легко отбрасывается, имея самый большой объем контура, который удовлетворяет заранее описанным предположениям.
def find_lungs(contours):
"""
Chooses the contours that correspond to the lungs and the body
First, we exclude non-closed sets-contours
Then we assume some min area and volume to exclude small contours
Then the body is excluded as the highest volume closed set
The remaining areas correspond to the lungs
Args:
contours: all the detected contours
Returns: contours that correspond to the lung area
"""
body_and_lung_contours = ()
vol_contours = ()
for contour in contours:
hull = ConvexHull(contour)
if hull.volume > 2000 and set_is_closed(contour):
body_and_lung_contours.append(contour)
vol_contours.append(hull.volume)
if len(body_and_lung_contours) == 2:
return body_and_lung_contours
elif len(body_and_lung_contours) > 2:
vol_contours, body_and_lung_contours = (list(t) for t in
zip(*sorted(zip(vol_contours, body_and_lung_contours))))
body_and_lung_contours.pop(-1)
return body_and_lung_contours
В качестве крайнего случая я показываю, что алгоритм не ограничивается только двумя областями легких. Осмотрите синий контур ниже:
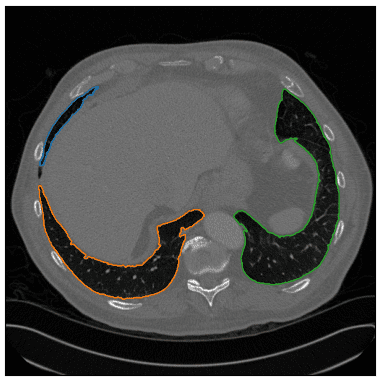
Изображение автора
Шаг 5: Преобразование контура в бинарную маску
Затем мы сохраняем его как отличный файл, поэтому нам нужно преобразовать набор точек в двоичную маску легких. Для этого я использовал библиотеку Python Pillow, которая рисует многоугольник и создает маску бинарного изображения. Затем сливаю все маски уже найденных контуров легких.
import numpy as np
from PIL import Image, ImageDraw
def create_mask_from_polygon(image, contours):
"""
Creates a binary mask with the dimensions of the image and
converts the list of polygon-contours to binary masks and merges them together
Args:
image: the image that the contours refer to
contours: list of contours
Returns:
"""
lung_mask = np.array(Image.new('L', image.shape, 0))
for contour in contours:
x = contour(:, 0)
y = contour(:, 1)
polygon_tuple = list(zip(x, y))
img = Image.new('L', image.shape, 0)
ImageDraw.Draw(img).polygon(polygon_tuple, outline=0, fill=1)
mask = np.array(img)
lung_mask += mask
lung_mask(lung_mask > 1) = 1
return lung_mask.T
Желаемая площадь легких в это просто количество ненулевых элементов, умноженное на два размера пикселя соответствующего изображения.
Области легких сохраняются в файле csv вместе с именем изображения.
Наконец, чтобы сохранить маску как изящную, я использовал значение 255 для области легких вместо 1, чтобы можно было отобразить ее в изящном средстве просмотра. Кроме того, я сохраняю изображение с аффинным преобразованием исходного среза КТ, чтобы его можно было отображать осмысленно (выровнять без каких-либо конфликтов вращения).
def save_nifty(img_np, name, affine):
"""
binary masks should be converted to 255 so it can be displayed in a nii viewer
we pass the affine of the initial image to make sure it exits in the same
image coordinate space
Args:
img_np: the binary mask
name: output name
affine: 4x4 np array
Returns:
"""
img_np(img_np == 1) = 255
ni_img = nib.Nifti1Image(img_np, affine)
nib.save(ni_img, name + '.nii.gz')
Наконец, я открыл маску с помощью обычного отличного средства просмотра для Linux, чтобы убедиться, что все прошло нормально. Вот снимки для среза номер 4:

Изображение автора
Я использовал бесплатную программу просмотра медицинских изображений под названием Ализа на Линукс.
Сегментируйте магистральные сосуды и вычислите отношение площади сосудов к площади легких.
Если внутри области легкого находится пиксель со значением интенсивности более -500 HU, то мы будем считать его сосудом.
Во-первых, мы делаем поэлементное умножение между КТ-изображением и маской легких, чтобы получить только легкие. После этого мы устанавливаем нули, полученные в результате поэлементного умножения, на -1000 (AIR в HU) и, наконец, оставляем в качестве сосудов только интенсивности, превышающие -500.
def create_vessel_mask(lung_mask, ct_numpy, denoise=False):
vessels = lung_mask * ct_numpy
vessels(vessels == 0) = -1000
vessels(vessels >= -500) = 1
vessels(vessels < -500) = 0
show_slice(vessels)
if denoise:
return denoise_vessels(lungs_contour, vessels)
show_slice(vessels)
return vessels
Один из примеров этого процесса можно проиллюстрировать ниже:
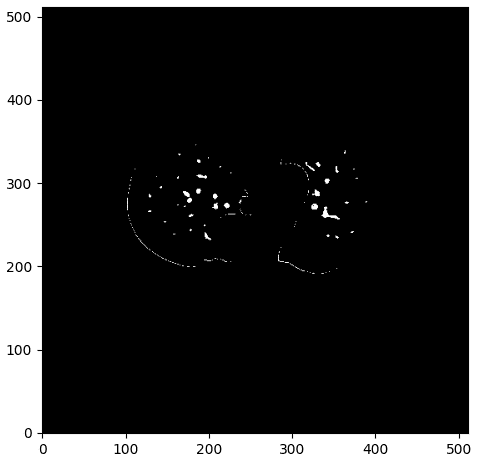
Маска сосуда с некоторым шумом. Изображение автора.
Анализ и улучшение результата сегментации
Как видите, у нас есть некоторые части контура легких, которых, как мне кажется, мы хотели бы избежать. С этой целью я создал функция шумоподавления который учитывает расстояние от маски до всех точек контура. Если он ниже 0,1, я устанавливаю значение пикселя в 0 и в результате исключаю их из обнаруженных сосудов.
def denoise_vessels(lung_contour, vessels):
vessels_coords_x, vessels_coords_y = np.nonzero(vessels)
for contour in lung_contour:
x_points, y_points = contour(:, 0), contour(:, 1)
for (coord_x, coord_y) in zip(vessels_coords_x, vessels_coords_y):
for (x, y) in zip(x_points, y_points):
d = euclidean_dist(x - coord_x, y - coord_y)
if d <= 0.1:
vessels(coord_x, coord_y) = 0
return vessels
Ниже вы можете увидеть разницу между изображением с шумоподавлением справа и исходной маской:
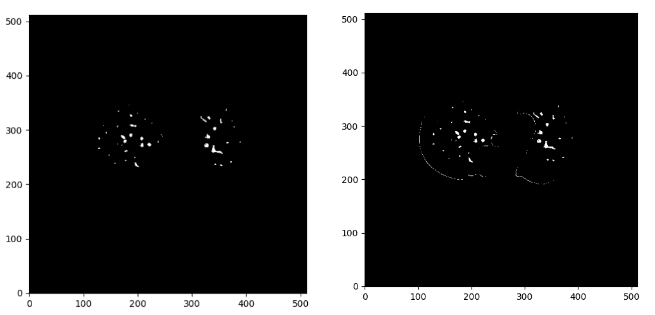
Изображение автора
Если мы наложим маску на исходное изображение КТ, мы получим:
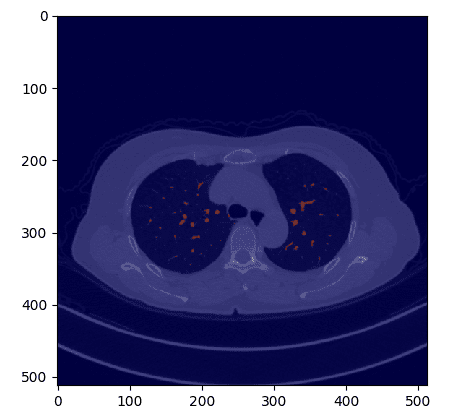
Изображение автора
def overlay_plot(im, mask):
plt.figure()
plt.imshow(im.T, 'gray', interpolation='none')
plt.imshow(mask.T, 'jet', interpolation='none', alpha=0.5)
Теперь, когда у нас есть маска, площадь сосуда вычисляется так же, как я делал для легких, с учетом размера отдельного пикселя изображения.
def compute_area(mask, pixdim):
"""
Computes the area (number of pixels) of a binary mask and multiplies the pixels
with the pixel dimension of the acquired CT image
Args:
lung_mask: binary lung mask
pixdim: list or tuple with two values
Returns: the lung area in mm^2
"""
mask(mask >= 1) = 1
lung_pixels = np.sum(mask)
return lung_pixels * pixdim(0) * pixdim(1)
Соотношения хранятся в файле csv в блокноте.
Заключение и дальнейшие чтения
Я считаю, что теперь у вас есть четкое представление о КТ-изображениях и их особенностях. Мы можем делать много удивительных вещей с такой богатой информацией о 3D-изображениях.
Поддержите нас, посмотрев наш проект на Гитхаб. Наконец, более продвинутые руководства см. в наших статьях по медицинской визуализации.
Меня попросили провести большой практический курс по искусственному интеллекту. Я думаю написать книгу по медицинской визуализации в 2021 году. До тех пор вы можете учиться на курсе Coursera. ИИ для медицины.
* Раскрытие информации: обратите внимание, что некоторые из приведенных выше ссылок могут быть партнерскими ссылками, и мы без дополнительных затрат для вас получим комиссию, если вы решите совершить покупку после перехода по ссылке.